Rから離れたくない人向けのDocker環境の操作: RStudio Serverを分析・開発の基盤にするために
この記事はRStudioアドベントカレンダーの21日目の記事です。もうすぐこのアドベントカレンダーも終わりですね。ハヤイ!
今年のはじめにこんな記事を書きました。
皆さんはDockerを利用していますでしょうか。今年のデータ分析系のアドベントカレンダーでもぞうさんがdockerが取り上げられています。
Rユーザの自分にとっては、Dirk EddelbuettelやCarl Boettigerなどが携わるrockerプロジェクトが整備されているのが嬉しいです。
rockerプロジェクトのdockerイメージの多くはRStudio Serverをイメージのベースとしており、お手軽にローカル環境とは別のRStudio環境が構築できます。また必要に応じて、rockerのdockerimageをベースに俺俺dockerimageへ拡張するのも簡単です。俺俺dockerimageの必要性についてはTokyo.RのLTで発表を行った過去の資料もあります。
そんな便利なdockerですが、dockerコマンドを覚えられなかったりコマンドラインから実行するのが面倒だったりします。特に自分は常にRStudioの画面を開いていたい側の人間なのでRからdockerコンテナやイメージの操作ができると良いです。良いです(大切なことなので二回言いました)。
... というわけでdockerパッケージを使います。これにより、Rから直接dockerのあれこれが行えるようになります(実態はPythonのdockerモジュールをreticulateパッケージでラップしているだけ)。大好きなRからできるので、dockerで何ができるかも理解しやすくて良いですね。
dockerパッケージ
以下の説明はdockerが利用できる環境で、dockerモジュールがインストールされていることを想定しています。あ、Rのdockerパッケージのインストールも忘れずに。CRANからインストールできます。まずはdockerを起動した状態で次のコードを実行します。Rスクリプトと一緒にコマンドラインで実行する際のdockerコマンドも示します。
# install.packages("docker") library(magrittr) library(docker) client <- docker$from_env()
インストールされているイメージ一覧を出力します。
# docker images client$images$list() # [[1]] # <Image: 'uribo/tiny_rocker_geospatial:latest'> # # [[2]] # <Image: 'rocker/tidyverse:latest'> # # [[3]] # <Image: 'kaggle/rstats:latest'> # # [[4]] # <Image: 'rocker/rstudio:latest'> # # [[5]] # <Image: 'rocker/r-base:latest'>
Dockerイメージをpullしてくるにはpull関数を使います。
# docker pull uribo/tiny_rocker_geospatial client$images$pull("uribo/tiny_rocker_geospatial")
利用するdockerイメージの用意ができたらコンテナを起動しましょう。次のRスクリプトの実行は、docker run --rm -p 8787:8787 uribo/tiny_rocker_geospatial と同じです。
rss_instance <- client$containers$run(image = "uribo/tiny_rocker_minimum", remove = TRUE, # RStudio上で操作を続ける際はTRUEにする detach = TRUE, ports = list("8787/tcp" = "8787")) rss_instance$start()
これでコンテナが起動しました。localhost:8787をブラウザで開きましょう。また次のコードでコンテナの一覧を確認してみると、きちんとコンテナが動いていることがわかります。
# docker ps -a client$containers$list() # [[1]] # <Container: a94576e6a2>
コンテナを終了するにはstop関数を使います。現在のコンテナはremoveオプションを有効にしていたため、コンテナを停止すると自動的にコンテナが削除されます。
# docker stop <コンテナID> rss_instance$stop() # 停止とともにコンテナは削除される # docker ps -a client$containers$list() # list
基本はこれでOKです。既存のコンテナを起動するには、まずコンテナのIDを知る必要があります。あるいはコンテナに名前をつけている場合にはそれを使うことも可能です。次はコンテナに名前をつけ、停止しても再起動できるように永続化させましょう。
rss_instance <- client$containers$run(image = "uribo/tiny_rocker_geospatial", remove = FALSE, detach = TRUE, name = "dev_1712", ports = list("8787/tcp" = "8787")) # 再起動を行うため停止します rss_instance$stop()
では停止しているコンテナを再起動します。再起動の方法には色々ありますが、ここではコンテナ名からコンテナidを抽出し、それを利用する方法を用います。
httr::http_error("http://localhost:8787") # Error in curl::curl_fetch_memory(url, handle = handle) : # Failed to connect to localhost port 8787: Connection refused
container_id <- client$containers$list(all = TRUE, filters = list("name" = "dev_1712")) %>% magrittr::extract2(1) %>% magrittr::use_series(id) cll <- docker$APIClient() cll$start(resource_id = container_id) httr::http_error("http://localhost:8787") # [1] FALSE
APIClient関数からcllというオブジェクトを作成し、コンテナを起動しました。dockerパッケージは便利ですが、コンテナの再起動をできないとRStudioを終了できないので困りますが、これで安心です。
明示的にコンテナを削除するには次のようにします。
cll$stop(resource_id = container_id) # docker rm <コンテナ ID> cll$remove_container(resource_id = container_id)
前述の通りdockerパッケージはPythonのdockerモジュールをラップしているので、dockerパッケージで何ができるのかを知りたい時はドキュメントを読むのが早いです。
ローカル環境のRStudioの設定を適用する
ここからは応用編です。起動したRStudio Serverでは、当然ながらRStudioの初期設定が適用されています。これではコンテナを作る度にぽちぽちとマウス操作によって設定をしていく必要が生じるので、楽にやりたいです。
なので、ローカルの設定をRStudio Serverへコピーするという方法をとっています。
system( paste0('docker cp /Users/uri/.rstudio-desktop/monitored/user-settings/user-settings ', container_id, ':/home/rstudio/.rstudio/monitored/user-settings/'))
APIがあるかと思ったのですが、なかなかうまくいかず...まだ試行錯誤なのですがこの方法で一応ローカルで動かしているRStudioのテーマやパネルの配置等の設定をRStudio Serverへ持ってくることができます。
注意としては、一度対象のコンテナを起動していないとコピーが成功しない(.rstudioという隠しフォルダが作成されない)、パスは絶対パスでなければいけないの2点です。
マウント: ローカルのファイルと同期させる
docker環境で作業して、それをローカルに保存したり、ローカルのコードをdocker環境で試す、ということがあります。それにはvolumeオプションを利用します。これはコンテナの作成時にやっておきます。
まずはdockerコンテナに構築したいローカルのパスとdockerコンテナ内の関係をオブジェクトとして作成します。modeは書き込みと保存ができるようにrwとしましょう。
mount <- list("/Users/uri/Documents/projects2016/jpmesh" = list("bind" = "/home/rstudio/jpmesh", # rw: read and write (ro: read only) "mode" = "rw")) rss_instance <- client$containers$run(image = "uribo/tiny_rocker_geospatial", remove = TRUE, detach = TRUE, volumes = mount, name = "dev_1712", ports = list("8787/tcp" = "8787"))

ユーザ、パスワードを変更する
RStudio Serverでは初期ユーザ名とパスワードがrstudioになっていますが、運用していくためには変更したいです。これもコンテナの起動時に設定しておきます。ここではenvironmentオプションを使います。USER、PASSWORDという値をそれぞれ任意のユーザ、パスワードにすることが可能です。以下は、ユーザ、パスワードの変更とついでに管理者ユーザとなるオプションも有効にする例です(追加でシステムのインストールが必要となることがあるため)。
rss_instance <- client$containers$run(image = "uribo/tiny_rocker_geospatial", remove = TRUE, detach = TRUE, environment = list("USER" = "piyo", "PASSWORD" = "hogepass", # sudo が有効になる "ROOT" = "TRUE"), ports = list("8787/tcp" = "8787"))

俺俺dockerfileの作成方法についてはまたの機会に。ちなみに、この記事で使っているコンテナは俺俺イメージの一つです。地理空間データを扱う上で欠かせない存在となったsfパッケージのインストールが面倒なのでサクッとできるようにしています。
https://hub.docker.com/r/uribo/tiny_rocker_geospatial/
Enjoy 🐳
2017年度版 RStudioを使ったReproducible Research、補足ポエム
この記事はRStudioアドベントカレンダーの10日目の投稿の補足です。私ももう、ゴールしても良いよね、という気になってきました(注)。本体は以前書いた記事で申し訳ないのですが、
になります。古くなったので刷新し、追記をしました。
RによるReproducible Researchの話は@psycle44さんによる5日目の記事でも書かれているのですが、補足として私の所感を述べたいと思います。以下はざっくりとした私の見解です。ポエムです。
ここで扱う、Reproducible Researchの話はRやPythonで記述されたコードにおける再現性の話です。海外では数年前から流れができ始め、現在では市民権を得て大きな流れになっているように感じます。一方日本国内でも少しずつですが認知されつつあり、一部のユーザの間ではかなり浸透しているという印象があります。
R Markdownは研究者ほど使うべき。ぞうさんがいうんだから間違いないですよ。
— kazutan v3.4.3 (@kazutan) June 19, 2016
そもそも再現性とは
学術論文では、調査地や調査対象、時期に始まり、データを得るための詳細や解析に利用したツール、ライブラリとそのバージョンまで書いたりしますよね。良い論文というのはしっかりとした再現性が担保できることだという話も聞いたことがあります。
で、現在ではRやPythonといったスクリプト言語で統計解析やモデリングを実行しますよね。そうなったら、そのコードで書いた内容も「方法」として扱っても良いよね、となるわけです。そうした意味ではバージョン管理をすることは一つの選択肢となり得ると思います。
Git?
Gitはバージョン管理システムの一つで、プログラマやエンジニアの中で使われてきたものですが最近ではデザイナや書籍の編集者も使っています。
Gitの恩恵はいろいろあり、その感じ方も多様ですが、Reproducible Researchでの再現性を保証するのに十分な機能を備えている気がします。Gitを用いる理由は自分自身のためでもあり、共同編集者のためでもあり、第三者のためでもあります。
以前、研究者に対してバージョン管理システムの話をしたことがありましたが、「Dropboxのように意識せずとも使えるようなものでないと使う気にならない」と言われました。Gitは確かにそこまで気がきくものではありません。ですが得られるものは大きいです。ぜひ一度使ってみてほしいものです。またGit以外で他に良いツールがあれば教えてほしいです。
何も全てのGitコマンドに精通する必要はありません。コミット、プッシュ、そしてブランチという概念について少しでもわかっていれば十分にGitを導入することができると思います。私も必要な操作については都度調べたりします。そしてRStudioではこうした最低限必要であろう機能が備わっています。逆に複雑なGitの機能は実装されていません(Shell機能を通して実行することはできます)。
まだRStudio or Gitを使ったことがないという人は今日できることから始めてみてはどうでしょう。まずはコミットで記録していくだけで良いと思います。
Happy Coding with R!
過去の資料とか
オススメ
中級者向けggplot2でこんな図が描きたい - 地図編
どーも。ggplot2は空手の一種として知られているので(要出典)普段の稽古が欠かせまん。今年を振り返り、ggplot2での作図について、いくつかの知見を共有します(書いている余裕がなかったんや...)
library(magrittr) library(jpndistrict)
## Loading required package: jpmesh
## This package provide map data is based on the Digital Map
## 25000(Map Image) published by Geospatial Information Authorityof
## Japan (Approval No.603FY2017 information usage
## <http://www.gsi.go.jp>)
library(sf)
## Linking to GEOS 3.6.1, GDAL 2.1.3, proj.4 4.9.3
library(tidyverse)
## ─ Attaching packages ───────────────── tidyverse 1.2.1 ─
## ✔ ggplot2 2.2.1.9000 ✔ purrr 0.2.4
## ✔ tibble 1.3.4 ✔ dplyr 0.7.4
## ✔ tidyr 0.7.2 ✔ stringr 1.2.0
## ✔ readr 1.1.1 ✔ forcats 0.2.0
## ─ Conflicts ─────────────────── tidyverse_conflicts() ─
## ✖ tidyr::extract() masks magrittr::extract()
## ✖ dplyr::filter() masks stats::filter()
## ✖ dplyr::lag() masks stats::lag()
## ✖ purrr::set_names() masks magrittr::set_names()
library(ggimage)
ggplot2で複数の図を一枚の図にまとめる方法あれこれ
図の中に小さな図(サブプロット)を配置する
こんな図を作りたい。

これは最近あった案件なのですが、やってみると結構手こずりました。また、作業のあとにggimageパッケージの更新があり、こちらの関数を利用するともっと楽になることが判明しました。残念。
自分がやった方法とggimageパッケージの利用例をそれぞれ示します。まずはオレオレから。
地図データは国土交通省 国土数値情報の提供する行政区域データ(国土地理院発行の数値地図(国土基本情報))となります。また、地図と合わせて掲載するデータを環境省 モニタリングサイト1000のページからデータをダウンロードしてきます。
# 北海道のShapefile 国土数値情報 # 行政区域データを利用 sf_pref01 <- jpndistrict::jpn_pref(1, district = FALSE) %>% st_simplify(dTolerance = 0.001)
まずはベースとなる地図を描画します。あとでグラフを追加したいので領域を広げておきます。
p_base <- sf_pref01 %>% ggplot() + geom_sf(fill = "white") + xlim(137, 149) + ylim(41, 46) + theme(panel.border = element_blank(), axis.title = element_blank(), axis.text = element_blank(), axis.ticks = element_blank(), plot.background = element_rect(colour = "black"), plot.caption = element_text(size = 6)) + labs(caption = "この地図は、国土地理院長の承認を得て、\n同院発行の数値地図(国土基本情報)電子国土基本図(地図情報)\nを使用したものである (承認番号 平28情使、第603号)")
今回は作図するのが主題なので、データの読み込み等の処理は説明を省略します。ダウンロードしたファイルの読み込みと整形、作図のための関数を書いておきます。
x <- list.files("~/Documents/projects2017/monit1000/data-raw/KOZ02/", pattern = ".xls$", full.names = TRUE) read_temp_xls <- function(path) { readxl::read_excel(path = path, sheet = 2, range = "A2:E10488", col_names = c("datetime", "above_temp", "below_5cm_temp", "below_10cm_temp", "notes"), col_types = c("guess", "text", "text", "text", "text"), skip = 1) %>% select(-below_5cm_temp, -notes) %>% gather(position, temperature, -datetime) %>% filter(!is.na(temperature)) %>% mutate(temperature = as.numeric(temperature)) } plot_temperature <- function(path, ...) { read_temp_xls(path) %>% ggplot(aes(datetime, temperature, color = position)) + geom_line() + ggtitle(...) + theme_set(theme_gray(base_size = 12, base_family = "IPAexGothic")) + theme_transparent() + theme(legend.position = "none", text = element_text(size = 6)) }
これらの関数を使って、一気に作図まで行います。このデータは、 モニタリングサイト1000の高山帯調査で記録された、大雪山の黒岳風衝地における地温・地表面温度です。凡例をつけていませんが、赤い線が地表、青い線が地下10cmでの気温(摂氏)を示しています。
plot_temperature(path = x[1], label = "黒岳風衝地")

ではこの2つの図を組み合わせて行きます。これにはggplotGrob()とannotation_custom()を利用します。
sub_p1 <- ggplotGrob(plot_temperature(path = x[1], label = "黒岳風衝地"))
p_base + annotation_custom(sub_p1, xmin = 136.2, xmax = 140.6, ymin = 43.4, ymax = 45.8)
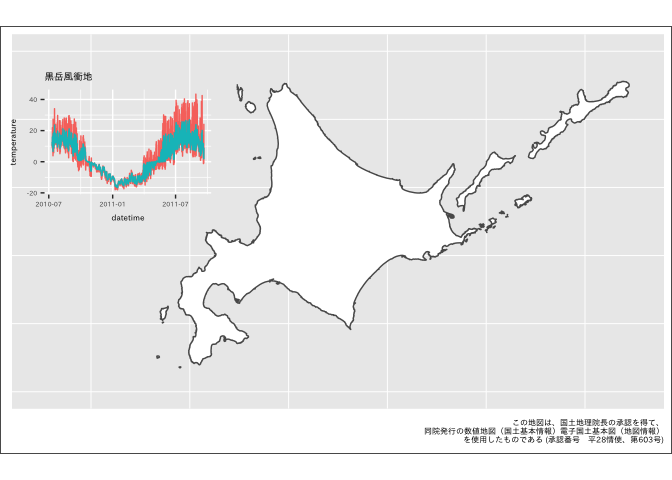
きちんと理解していないので適当ですが、このように、図中に含ませたい図をggplotGrob()でgtableオブジェクトに変換し、annotation_custom()で上乗せする、という方法です。annotation_custom()の引数で図の大きさ、配置を調整します。
で、これだと図が多くなると手間ですよね。というわけでggimageを使います。私が作業する時には更新が間に合いませんでしたが、今度からはこっちを使いたいと思います。簡単です。
まずは図を用意しましょう。今度は3地点の図を一度に載せます。グラフの描画にもpurrrは便利ですね。
plots <- map2(.x = x[c(1, 5, 9)], .y = c("黒岳風衝地", "黒岳石室", "赤岳第4雪渓"), .f = ~plot_temperature(path = .x, label = .y)) # 個別の図はリストに格納されています # plots[[1]]
ggimageのgeom_subview()はデータフレームに配置する座標や図を格納し他データフレームを展開します。そのためまずは図を配置する座標、サイズ、含めたい図のオブジェクトからなるデータフレームを作りましょう。
# plot変数にlistで図を格納する df_plots <- data_frame(x = c(138.2, 138.2, 146), y = c(45.4, 43.4, 41.8), width = 3.5, height = 1.7, plot = list(plots[[1]], plots[[2]], plots[[3]]))
これをgeom_subview()に渡します。
p_add <- p_base + geom_subview(aes(x = x, y = y, subview = plot, width = width, height = height), data = df_plots) p_add
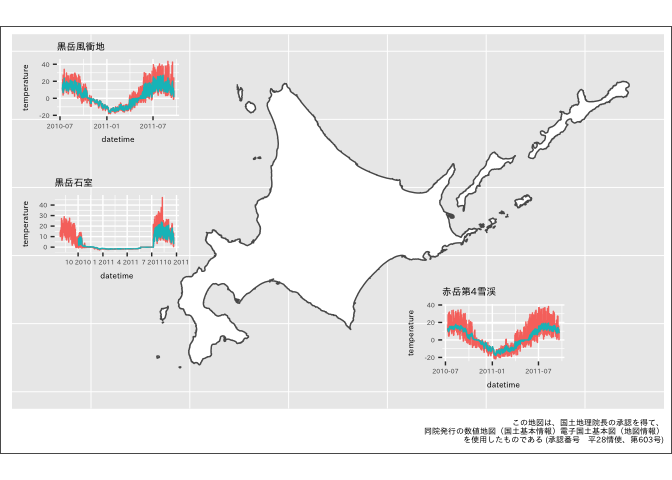
できたー!格好いい図が描けました。よかったですね。(軸が潰れてしまっていますが、一旦は無視してください)
出典: 「モニタリングサイト1000高山帯調査 地温・地表面温度調査」(環境省生物多様性センター) (http://www.biodic.go.jp/moni1000/findings/data/index_file_earth-temp.html)
この図におけるグラフデータは「モニタリングサイト1000高山帯調査 地温・地表面温度調査」(環境省生物多様性センター)を使用し、瓜生真也が作成・加工したものである。(http://www.biodic.go.jp/moni1000/findings/data/index_file_earth-temp.html)
沖縄を移動、南方諸島を省略した日本地図

さてもう一段。今日はRアドベントカレンダーなので2つ目のtipsです。
日本地図を描画する際、沖縄県や小笠原群島を中心とした南方諸島の島々の位置が気になることがあります。そのため地図の配置を変えるという試みがこれまでされてきているのですが、これのggplot2版というのは見たことがありません。
これもggimage::geom_subview()を使うとできてしまうのでやってみましょう。完成図は上に示したものです。
sf_ja <- 1:47 %>% magrittr::extract(-13) %>% map(~jpndistrict::jpn_pref(pref_code = ., district = FALSE)) %>% reduce(rbind) %>% st_simplify(dTolerance = 0.01) sf_pref13 <- jpn_pref(pref_code = 13, district = TRUE) %>% st_simplify(dTolerance = 0.01) %>% mutate(city_code = as.numeric(city_code)) %>% filter(city_code != 13421) %>% st_union() %>% as.data.frame() %>% mutate(jis_code = "13", prefecture = "東京都") %>% magrittr::set_names(c("geometry", "jis_code", "prefecture")) %>% st_as_sf() sf_ja_omit47 <- sf_ja %>% filter(jis_code != "47") sf_ja_pref47 <- sf_ja %>% filter(jis_code == "47") sf_ja_pref47$geometry %<>% magrittr::add(c(5.6, 17.5)) sf_ja_pref47 %<>% st_set_crs(value = 4326) p <- ggplot(sf_ja_omit47) + geom_sf() + geom_sf(data = sf_pref13, inherit.aes = TRUE) + geom_sf(data = sf_ja_pref47, inherit.aes = TRUE) + geom_segment(aes(x = round(st_bbox(sf_ja_omit47)[1], 0), xend = 132.5, y = 40, yend = 40)) + geom_segment(aes(x = 132.5, xend = 138, y = 40, yend = 42)) + geom_segment(aes(x = 138, xend = 138, y = 42, yend = round(st_bbox(sf_ja_omit47)[4], 0))) + xlab(NULL) + ylab(NULL) + theme(plot.caption = element_text(size = 6)) + labs(caption = "この地図は、国土地理院長の承認を得て、\n同院発行の数値地図(国土基本情報)電子国土基本図(地図情報)\nを使用したものである (承認番号 平28情使、第603号)") p
いいですね。Enjoy! 時間がなくてコードの説明が足りていないので、後ほど追記します。