データフレームの特徴をもっと早く掴みたい ~ ハドリーへの挑戦
探索的なデータ分析 (Explore Data Analysis: EDA)を行う際は、データの要約や欠損の有無の確認、可視化が欠かせない作業となります。
特に可視化は、データのもつ性質や関係を表現するのに大変役立ちます。一方で、可視化に用いた図はコードとは別に保存する必要があったり、作図のためのコードを書いたりと、面倒な側面もあります。
… きちんとした作図は面倒だけどデータの性質や欠損について把握したい。そんな時にはコンソール上での可視化を試しましょう。そのためのパッケージをHadley Wickhamが開発しています。
https://github.com/hadley/precis
Rにはそもそも、オブジェクトの情報を要約してくれるsummary()関数があるのですが、この precisパッケージは、それを置き換えるような設計を目指しているそうです。早速使ってみましょう。
# githubinstall::githubinstall('precis') library(precis)
現在precisが提供するのは、それぞれベクトルとデータフレームの要約を行う2つの関数、precis_v()とprecis()です。precis()が返す個々の情報は、precis_v()によるものです。
データフレームにprecis()を実行すると、次のような出力が行われます。summary()と異なり、返り値もデータフレームであるのが特徴で、データフレームの変数名を示すname、変数のデータ型を示すtype、そして変数をベクトルとして評価した時の要約した情報がprecis列に格納されています。
precis(iris)
## # data.frame [150 x 5]
## name type precis
## <chr> <chr> <chr>
## 1 Sepal.Length dbl 4.3 [ 5.1 ( 5.8) 6.4] 7.9
## 2 Sepal.Width dbl 2.0 [ 2.8 ( 3.0) 3.3] 4.4
## 3 Petal.Length dbl 1.00 [ 1.60 ( 4.35) 5.10] 6.90
## 4 Petal.Width dbl 0.1 [ 0.3 ( 1.3) 1.8] 2.5
## 5 Species fctr setosa (50) versicolor (50) virginica (50)
precis(mtcars)
## # data.frame [32 x 11]
## name type precis
## <chr> <chr> <chr>
## 1 mpg dbl 10.4 [ 15.4 ( 19.2) 22.8] 33.9
## 2 cyl dbl 4 (11) 6 (7) 8 (14)
## 3 disp dbl 71.1 [121.0 (196.0) 334.0] 472.0
## 4 hp dbl 52 [ 96 ( 123) 180] 335
## 5 drat dbl 2.76 [ 3.08 ( 3.70) 3.92] 4.93
## 6 wt dbl 1.51 [ 2.54 ( 3.32) 3.65] 5.42
## 7 qsec dbl 14.5 [ 16.9 ( 17.7) 18.9] 22.9
## 8 vs dbl 0 (18) 1 (14)
## 9 am dbl 0 (19) 1 (13)
## 10 gear dbl 3 (15) 4 (12) 5 (5)
## 11 carb dbl 1 [ 2 ( 2) 4] 8
precis列の値は、対象の変数のデータ型に応じて異なります。例えば数値であれば、最小値、第一四分位、中央値、第三四分位、最大値の値です(summary()で得られる情報と同じ)。最大値と最小値の間が[括弧になっており、中央値については(括弧が使われているのでわかりやすいですね。また、因子型や文字列型については、順位、カウント値の情報が出力されます。
precis()の良い点は、これに加えて引数histogramでTRUEを指定した際に、数値データのヒストグラムをコンソール上で描画してくれるところです。例えば次の例では、hist(iris$Sepal.Length)とせずともデータの分布が掴めるので、偏りがあるとか正規分布に近い、といったことが把握しやすいという利点になります。
precis(iris, histogram = TRUE)
## # data.frame [150 x 5]
## name type precis
## <chr> <chr> <chr>
## 1 Sepal.Length dbl 4.3 ▂▂▃▇▇▃▇▇▃▅▇▃▅▂▂▁▁▂▁ 7.9
## 2 Sepal.Width dbl 2.0 ▁▁▂▅▇▅▃▂▂▁▁▁ 4.4
## 3 Petal.Length dbl 1.0 ▇▂▁▁▁▂▃▃▃▃▁▁ 6.9
## 4 Petal.Width dbl 0.1 ▇▃▁▁▂▂▅▃▃▂▂▂▁ 2.5
## 5 Species fctr setosa (50) versicolor (50) virginica (50)
… でも、それだけでなく、欠損値やカウントの数を知りたいんだ!という気持ちが改造したのが次のuncover()関数になります。gistからソースコードを引っ張って来ることで利用可能になります。
devtools::source_gist("f275260bbd1da5f613241458c43adb2d", filename = "uncover.R")
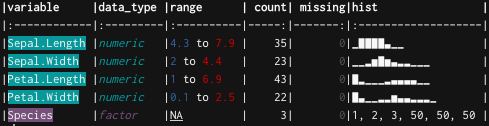
uncover(mtcars)
### A tibble: 11 x 6
## variable data_type range count missing hist
## <chr> <chr> <chr> <int> <int> <chr>
## 1 mpg numeric 10.4 to 33.9 25 0 ▂▁▇▃▅▅▂▂▁▁▂▂
## 2 cyl numeric 4 to 8 3 0 ▅▁▁▃▁▁▁▇
## 3 disp numeric 71.1 to 472 27 0 ▅▇▃▁▃▃▃▁▂
## 4 hp numeric 52 to 335 22 0 ▇▇▃▃▁▁
## 5 drat numeric 2.76 to 4.93 22 0 ▂▂▇▂▁▅▇▃▂▁▁▁
## 6 wt numeric 1.513 to 5.424 29 0 ▃▃▃▇▅▁▁▂
## 7 qsec numeric 14.5 to 22.9 30 0 ▁▂▂▇▃▂▁▁▁
## 8 vs numeric 0 to 1 2 0 ▇▁▁▁▁▁▁▁▁▅
## 9 am numeric 0 to 1 2 0 ▇▁▁▁▁▁▁▁▁▅
##10 gear numeric 3 to 5 3 0 ▇▁▁▁▅▁▁▁▁▂
##11 carb numeric 1 to 8 6 0 ▅▇▁▂▁▇▁▁▁▁▁▁▁▁
uncover(ggplot2::diamonds)
### A tibble: 10 x 6
## variable data_type range count missing hist
## <chr> <chr> <chr> <int> <int> <list>
## 1 carat numeric 0.2 to 5.01 273 0 <chr [1]>
## 2 cut ordered_factor Fair to Ideal 5 0 <data.frame [5 x 2]>
## 3 color ordered_factor D to J 7 0 <data.frame [7 x 2]>
## 4 clarity ordered_factor I1 to IF 8 0 <data.frame [8 x 2]>
## 5 depth numeric 43 to 79 184 0 <chr [1]>
## 6 table numeric 43 to 95 127 0 <chr [1]>
## 7 price integer 326 to 18823 11602 0 <chr [1]>
## 8 x numeric 0 to 10.74 554 0 <chr [1]>
## 9 y numeric 0 to 58.9 552 0 <chr [1]>
##10 z numeric 0 to 31.8 375 0 <chr [1]>
range列はprecis()同様、変数のデータの幅を示すものですが、こちらは最小値と最大値しかわかりません。順位のある因子型は順位の並びで最小・最大のものが出力されるようになっています。
また、変数が数値であればprecis()と同じくヒストグラムを出力します。が、因子型や文字列が含まれるデータでは次のようにhist列の情報はリストカラムとして格納されます。これを取り出すには、dplyr::filterでデータ型の制限をし、tidyr::unnest()でリストカラムを解除してください。
uncover(ggplot2::diamonds) %>% dplyr::filter(data_type %in% c("numeric", "integer")) %>% tidyr::unnest()
### A tibble: 7 x 6
## variable data_type range count missing hist
## <chr> <chr> <chr> <int> <int> <chr>
##1 carat numeric 0.2 to 5.01 273 0 ▇▇▅▁▁▁▁▁▁▁▁
##2 depth numeric 43 to 79 184 0 ▁▁▁▁▇▁▁▁
##3 table numeric 43 to 95 127 0 ▁▁▂▇▁▁▁▁▁▁▁
##4 price integer 326 to 18823 11602 0 ▇▃▂▁▁▁▁▁▁▁
##5 x numeric 0 to 10.74 554 0 ▁▁▁▁▇▇▇▂▁▁▁
##6 y numeric 0 to 58.9 552 0 ▃▇▁▁▁▁▁▁▁▁▁▁
##7 z numeric 0 to 31.8 375 0 ▇▁▁▁▁▁▁
uncover(ggplot2::diamonds) %>% dplyr::filter(data_type %in% c("ordered_factor")) %>% tidyr::unnest()
### A tibble: 20 x 7
## variable data_type range count missing value.Var1 value.Freq
## <chr> <chr> <chr> <int> <int> <chr> <int>
## 1 cut ordered_factor Fair to Ideal 5 0 Fair 1610
## 2 cut ordered_factor Fair to Ideal 5 0 Good 4906
## 3 cut ordered_factor Fair to Ideal 5 0 Very Good 12082
## 4 cut ordered_factor Fair to Ideal 5 0 Premium 13791
## 5 cut ordered_factor Fair to Ideal 5 0 Ideal 21551
## 6 color ordered_factor D to J 7 0 D 6775
## 7 color ordered_factor D to J 7 0 E 9797
## 8 color ordered_factor D to J 7 0 F 9542
## 9 color ordered_factor D to J 7 0 G 11292
##10 color ordered_factor D to J 7 0 H 8304
##11 color ordered_factor D to J 7 0 I 5422
##12 color ordered_factor D to J 7 0 J 2808
##13 clarity ordered_factor I1 to IF 8 0 I1 741
##14 clarity ordered_factor I1 to IF 8 0 SI2 9194
##15 clarity ordered_factor I1 to IF 8 0 SI1 13065
##16 clarity ordered_factor I1 to IF 8 0 VS2 12258
##17 clarity ordered_factor I1 to IF 8 0 VS1 8171
##18 clarity ordered_factor I1 to IF 8 0 VVS2 5066
##19 clarity ordered_factor I1 to IF 8 0 VVS1 3655
##20 clarity ordered_factor I1 to IF 8 0 IF 1790
uncover(datasets::Titanic) %>% dplyr::filter(data_type %in% c("numeric", "integer")) %>% tidyr::unnest()
### A tibble: 1 x 6
## variable data_type range count missing hist
## <chr> <chr> <chr> <int> <int> <chr>
##1 n numeric 0 to 670 22 0 ▇▂▁▁▁▁▁▁▁▁▁▁▁▁
コードを書いてみて思ったのが、precis::precis()でいいじゃん…ということでした。いやでもuncover()も役立つ時があるはず!(ハドリーへの挑戦と思ってこの思い出は胸にしまっておきます)
いやあ、欲を言えばこういうことがしたいんですよね(次回に続く…)
RStudioを使ってKaggleコンペティションをやっていくためのプロジェクトテンプレートを作っています
はじめに注意書きですが、この記事で書かれているプロジェクトテンプレート機能は、今日現在、開発版のRStudioを利用したものです。厳密には、実行のためにはv1.1.28以上である必要があります。おそらく次の安定版には盛り込まれる機能であると思うので、しばしお待ちください。
というわけで、タイトルにある通り、Kaggleコンペ向けのRStudioのプロジェクトテンプレートを作りました。プロジェクトテンプレートって何? どういうのを作ったの? という話をします。
プロジェクトテンプレート
RStudioでは、プロジェクト機能により複数の作業環境を構築することが可能です。プロジェクトテンプレートとは、ユーザが定義したプロジェクトをテンプレートして利用可能にしたものです。今までもRパッケージのためにプロジェクトを作成する場合には、パッケージ開発に必要な構造をもったプロジェクトを生成するテンプレートが利用されていました。新しい機能ではそれ以外にもRcppを使ったパッケージ開発や、ユーザの作ったプロジェクトテンプレートが利用できるようになっています。
kagglepro
kaggleproというのが、作ったパッケージの名前です。GitHubで公開しています。
Kaggleのようなコンペティションでは、提供されるデータを保存するディレクトリ、モデルを書くディレクトリ、可視化のための作図を行うディレクトリ、提出するファイルをモデルごとに出力したディレクトリなど、コンペティションの種類を問わず、いくつか共通の構造を持ったディレクトリを構成することがしばしばあります。
テンプレートとして、空のフォルダを用意しておいてそれをコピペしても良いのですが、せっかくなので、コンペでよく使うフォルダ構成をプロジェクト立ち上げ時に生成してしまおう、というのがkaggleproの開発の動機となっています。
またKaggleでは、Kaggle Kernelsと呼ばれる (以前はScriptsという名前だった)、スクリプト実行環境および公開の場が設けられています。そこではDockerを使った環境が用意されており、Kaggleが用意するKernelで利用するDockerイメージを元に手前でDockerfileを作っておけば、ローカルでもKernelと同じ環境を再現することが可能です。そのためkaggleproでも、KaggleのRユーザ向けのDockerイメージをベースとしたDockerfileの生成を行えるようにしました。既存のDockerfileがある場合、それをインポートすることも可能です。
kaggleproの使い方です。GitHubからパッケージをインストールしてください(冒頭にも述べたように、kaggleproを利用するには開発版のRStudio(v1.1.28以上)が必要です。)。
# GitHubリポジトリからのパッケージインストールを実行する # devtoolsパッケージがインストールされていない場合 # install.packages("devtools") devtools::install_github("uribo/kagglepro")
R操作の中でkaggleproを利用することはありません。RStudioのプロジェクトを新規作成する時にのみこのパッケージを使います。次の画像にも示すように、RStudioのメニューバーから「File」、「New Project…」を選択し、「New Directory」から「Kaggle Competition」というのを見つけて押します。次に表示される画面は、プロジェクトを生成するディレクトリとDockerfileに関する情報を入力するものです。ここの入力は適当に行ってください。

「Create Project」ボタンを押すと、プロジェクトが作成されます。kaggleproでは現在、次のようなディレクトリ構成のプロジェクトを作ります。
dockerfile *.Rproj data/ input/ source/ submission/
ちなみにですがkaggleproという名前にはKaggle向けのProjectテンプレート、という意味とPro Kaggle User (Kaggle Grandmaster)という2つの意味が込められています(本当か?)Kaggleの経験が浅く、どのようなディレクトリがあれば十分かという点にまだ不安が残りますが、よければこのオレオレテンプレートを使ってKaggleコンペに挑んでみてください。こういう風にした方が良いなどの改善や要望がありましたらプルリクエストを送っていただけると嬉しいです。
Enjoy!
Rおじさん、Pythonistaになる
こちらをご覧ください。踏み絵ではありません。R上で地理空間データを扱うPythonモジュール、geopandasによる作図を行なっている画面です。

え、RでPythonを!?と驚かれる方もいるかもしれませんが、reticulateというRパッケージを使うことで、ほぼストレスフリーでPythonのモジュールや関数がR上で利用可能になります。先の図は次のコードによって実行されました。
library(reticulate)
# モジュールの呼び出し gpd <- import("geopandas") plt <- import("matplotlib.pyplot") # サンプルデータの読み込み world <- gpd$read_file(gpd$datasets$get_path("naturalearth_lowres")) # データセットの確認 head(world) # ではないので注意 world$head(n = 3L) # 作図と出力 world$plot() plt$show()
reticulateはRStudio社が中心となり開発されている、Pythonのモジュール、クラス、関数をRで利用可能にするRのパッケージです。
R Interface to Python • reticulate
先ほどのコードを振り返りながら、reticulateでできることを見て行きましょう。参考のためPythonでのコードを併記します。
import geopandas as gpd world = gpd.read_file(gpd.datasets.get_path('naturalearth_lowres')) world.head(3) world.plot()
Rで言う所のライブラリ(パッケージ)、Pythonモジュールはimport()関数を使って呼び出します。Pythonと同じ関数になっているのでわかりやすいですね。
gpd <- import("geopandas") # Rのclass()でクラスを確認。Pythonモジュールとして扱われている class(gpd) # [1] 'python.builtin.module' # 'python.builtin.object'
読み込んだモジュールの関数を使うにはモジュールオブジェクト中で$演算子を使って参照します。これによりgeopandasモジュールのサンプルデータをオブジェクトとして利用可能にします。次のコードの最初2行を見る通り、Pythonの.をRでの参照機能をもつ$演算子に置換しているだけです。これはとてもわかりやすいと思います。
world <- gpd$read_file(gpd$datasets$get_path("naturalearth_lowres")) world$head(n = 3L) # continent gdp_md_est geometry \ 0 Asia # 22270.0 POLYGON ((61.21081709172574 # 35.65007233330923,... 1 Africa # 110300.0 (POLYGON ((16.32652835456705 # -5.87747039146621... 2 Europe 21810.0 # POLYGON ((20.59024743010491 # 41.85540416113361,... iso_a3 name # pop_est 0 AFG Afghanistan 28400000.0 1 # AGO Angola 12799293.0 2 ALB Albania # 3639453.0 # パイプ演算子だって使えちゃう world <- # gpd$datasets$get_path('naturalearth_lowres') # %>% gpd$read_file() world$tail(n = 3L)
関数内の引数はR同様、括弧の中で引数名と値の組み合わせを指定します。
world$plot()
普段慣れている$演算子による操作が行えるので、Pythonも怖くないですね。さらにRStudioを使っていると下の図のように、入力補完やドキュメントの内容(引数含めて)を表示してくれるので、Pythonに慣れていない私のような人間でも安心ですね。学習が捗りそうです。

しっかりとドキュメントを読みたいときは、py_help()を使います。ここでもRStudioを使っていると、ドキュメントの参照が手軽で良いですね。
py_help(world$tail)
Python環境の切り替え
Pythonを使っていて悩ましい問題の一つが、2系と3系を始めとした複数環境の切り替えです。reticulateではuse_python()を始めとして、condaや仮想環境下のdocker(bhaskarvkさんの野心的なプロジェクトを参照)でのPythonも実行できるようになっています。例を見てみましょう。
実際のPython環境の切り替えは、Rのセッション中にimport()が実行されたタイミングで行われます。そのため次のコードは一度Rセッションを終了してから再度実行しているものになります。
sys <- import("sys") sys$version # [1] '3.6.1 |Anaconda 4.4.0 (x86_64)| # (default, May 11 2017, 13:04:09) # \n[GCC 4.2.1 Compatible Apple LLVM 6.0 # (clang-600.0.57)]' library(reticulate) use_python("/usr/bin/python") sys <- import("sys") sys$version # [1] 2.7.13 (default, Jul 15 2017, # 12:14:18) \n[GCC 4.2.1 Compatible # Apple LLVM 8.1.0 (clang-802.0.42)] library(reticulate) use_condaenv("py36con", conda = "/Users/uri/.pyenv/versions/anaconda3-4.4.0/bin/conda", required = TRUE) sys <- import("sys") sys$version # [1] '3.6.1 |Anaconda 4.4.0 (x86_64)| # (default, May 11 2017, 13:04:09) # \n[GCC 4.2.1 Compatible Apple LLVM 6.0 # (clang-600.0.57)]'
どの環境を利用するかには優先順位があるようで、use_*()により指定された環境、Sys.setenv(RETICULATE_PYTHON)でのパス、Sys.which('python')により見つかるパス、慣用的にPythonが配置されるパスだそうです。都度use_*()を実行するのも手間なので、Sys.setenv()に値を記述しておくと良いかもしれません。なお、RStudioでのSys.which('python')の参照先は2系になるらしいので注意です(それでハマった)。
その他にも
reticulateパッケージの良いところは他にもあって、
が代表的な機能です。
RodeoやらPyCharmやら色々とPythonの開発環境を試しましたが、RStudioに慣れすぎていてRStudio依存になってしまっているのでreticulate、すごく助かります。
より詳しいことはRStudioがvignettesを書いているのでそちらを見ていただきたいのですが、nakamichiさんが翻訳してくださっているのでそちらも是非!ちなみにですがnakamichiさんは他にもたくさんのvignettesを多数翻訳していらっしゃいます。
少しずつですが、reticulateによるPythonの利用を前提にしたRパッケージも増えてきました (例 docker, tensorflow, , RQGIS)。今後ますます、RとPythonの連携による実現領域が広がっていくことが期待できますね。Pythonとのインターフェイスを用意してくれるRStudio最高!PythonとR仲良くしようね!
Enjoy!!