zipangu v0.3.0をCRANにリリースしました
日本人が扱う住所や年号、漢数字のデータ操作を楽にするRパッケージ
{zipangu}の最新バージョン
v0.3.0をCRANにリリースしました。前回のリリースに引き続き、多くの方が開発に協力してくださいました。ありがとうございます。この記事では新たに追加された機能や改善された点について紹介します。
新バージョンの{zipangu}は次のコマンドを実行することでインストールされます。
install.packages("zipangu") library(zipangu) rlang::is_installed("zipangu", version = "0.3.0")
それでは新機能の紹介に入ります。
新機能
str_jnormalize()による文字列正規化
以前から日本語文字列の正規化の関数としてstr_jconv()があり、全角文字列と半角文字列の変換が可能でしたが、今回追加されたstr_jnormalize()により、文字列正規化の機能が強化されました。
str_jnormalize()は解析前に行うことが望ましい文字列の正規化処理として定義されているneologdの処理内容を実現するものです。例をあげると「1回以上連続する長音記号は1回に置換」などがあります。対象の文字列を引数に与えて実行します。
str_jnormalize("スーーーパーマン") ## [1] "スーパーマン"
日本語文字列の前処理としてルールを統一し、一元的に実行できるようになっているので便利だと思います。 こちらの関数は@paithiov909さんの貢献により実装されました。ありがとうございます。
平仮名と片仮名のベクトルを生成
Rに定数として用意されているLETTERSを使ったことがある人は多いと思います。これはアルファベットを格納した文字列のオブジェクトです。今回、日本語ユーザのlettersとして新たにkana()を用意しました。こちらはlettersとは異なり関数として機能しますが、歴史的仮名遣を含めた平仮名と片仮名を出力します。
kana("hira") ## [1] "あ" "い" "う" "え" "お" "か" "き" "く" "け" "こ" "さ" "し" "す" "せ" "そ" ## [16] "た" "ち" "つ" "て" "と" "な" "に" "ぬ" "ね" "の" "は" "ひ" "ふ" "へ" "ほ" ## [31] "ま" "み" "む" "め" "も" "や" "ゆ" "よ" "ら" "り" "る" "れ" "ろ" "わ" "を" ## [46] "ん" kana("kata") ## [1] "ア" "イ" "ウ" "エ" "オ" "カ" "キ" "ク" "ケ" "コ" "サ" "シ" "ス" "セ" "ソ" ## [16] "タ" "チ" "ツ" "テ" "ト" "ナ" "ニ" "ヌ" "ネ" "ノ" "ハ" "ヒ" "フ" "ヘ" "ホ" ## [31] "マ" "ミ" "ム" "メ" "モ" "ヤ" "ユ" "ヨ" "ラ" "リ" "ル" "レ" "ロ" "ワ" "ヲ" ## [46] "ン"
kana()は第一引数typeによって出力する文字列の種類を指定します。ここではhira、hiraganaが平仮名、kataおよびkatakanaが片仮名の指定となります。
デフォルトでは五十音図の清音「あ」から「ん」が出力されますが、「が」や「だ」などの濁音、「ぱ」「ぽ」といった半濁音、「ぁ」などの捨て仮名(小書き)、さらに歴史的仮名遣で使われる「ゐ」や「ゑ」を含める、またはこれらに限定するおオプションを用意しました。
# 濁音を含めて出力 kana("hiragana", dakuon = TRUE) ## [1] "あ" "い" "う" "え" "お" "か" "が" "き" "ぎ" "く" "ぐ" "け" "げ" "こ" "ご" ## [16] "さ" "ざ" "し" "じ" "す" "ず" "せ" "ぜ" "そ" "ぞ" "た" "だ" "ち" "ぢ" "つ" ## [31] "づ" "て" "で" "と" "ど" "な" "に" "ぬ" "ね" "の" "は" "ば" "ひ" "び" "ふ" ## [46] "ぶ" "へ" "べ" "ほ" "ぼ" "ま" "み" "む" "め" "も" "や" "ゆ" "よ" "ら" "り" ## [61] "る" "れ" "ろ" "わ" "を" "ん" "ゔ" # 半濁音のみの出力 kana("hiragana", handakuon = TRUE, core = FALSE) ## [1] "ぱ" "ぴ" "ぷ" "ぺ" "ぽ" # 歴史的仮名遣 kana("kata", historical = TRUE) ## [1] "ア" "イ" "ウ" "エ" "オ" "カ" "キ" "ク" "ケ" "コ" "サ" "シ" "ス" "セ" "ソ" ## [16] "タ" "チ" "ツ" "テ" "ト" "ナ" "ニ" "ヌ" "ネ" "ノ" "ハ" "ヒ" "フ" "ヘ" "ホ" ## [31] "マ" "ミ" "ム" "メ" "モ" "ヤ" "ユ" "ヨ" "ラ" "リ" "ル" "レ" "ロ" "ワ" "ヰ" ## [46] "ヱ" "ヲ" "ン"
type引数を指定せずに、関数名で平仮名片仮名の指定が可能なhiragana()、katakana()もあります。
hiragana() ## [1] "あ" "い" "う" "え" "お" "か" "き" "く" "け" "こ" "さ" "し" "す" "せ" "そ" ## [16] "た" "ち" "つ" "て" "と" "な" "に" "ぬ" "ね" "の" "は" "ひ" "ふ" "へ" "ほ" ## [31] "ま" "み" "む" "め" "も" "や" "ゆ" "よ" "ら" "り" "る" "れ" "ろ" "わ" "を" ## [46] "ん" katakana() ## [1] "ア" "イ" "ウ" "エ" "オ" "カ" "キ" "ク" "ケ" "コ" "サ" "シ" "ス" "セ" "ソ" ## [16] "タ" "チ" "ツ" "テ" "ト" "ナ" "ニ" "ヌ" "ネ" "ノ" "ハ" "ヒ" "フ" "ヘ" "ホ" ## [31] "マ" "ミ" "ム" "メ" "モ" "ヤ" "ユ" "ヨ" "ラ" "リ" "ル" "レ" "ロ" "ワ" "ヲ" ## [46] "ン"
ダミーデータを作る時など、いちいち文字を入力する必要がなくなるので便利です。
都道府県名の正規化
都道府県名の表記揺れ、例えば「東京」や「茨城」といった都道府県名の最後の県などの表記がないものと「徳島県」といった最後の表記が含まれるものが混在する場合に「東京都」や「茨城県」に修正する処理を実現する関数をharmonize_prefecture_name()として用意しました。末尾の「県」を追加するだけでなく、反対に「県」の除外もオプションで指定可能です。
harmonize_prefecture_name(c("東京", "北海道", "沖縄"), to = "long") ## [1] "東京都" "北海道" "沖縄県" harmonize_prefecture_name(c("東京都", "北海道", "沖縄県"), to = "short") ## [1] "東京" "北海道" "沖縄"
改善
祝日評価のための関数についての変更が多くありました。既存のバージョンで挙動がおかしかった問題も解決されました。以下、要点です。
おわりに
今回のバージョンも多くの方の協力により機能追加・修正を行うことができました。ありがとうございます! zipanguでは引き続き、新機能の提案や問題点の報告、使用感についてのご意見を募集しています。 気になる点がありましたら気兼ねなくTwitter、GitHub等でコメントください。
Enjoy!
改訂2版 RユーザのためのRStudio実践入門のおすすめポイント
「RユーザのためのRStudio[実践]入門」の改訂2版を執筆者の一人、湯谷さん(id:yutannihilation)から頂いた。ありがとうございます。感謝の言葉と合わせて、書籍の紹介と読んだ感想について書いておく。
電子版はすでに発売されていて、紙版も今月3日に発売ということだ。この記事を読む人がRに対してどの程度慣れているか、また初版を持っているかどうかわからないのでそれぞれの視点で参考になるように書いてみた。購入を迷われている方の参考となれば幸いである。
R言語の初心者、初版を持っていない方へ
本書は2018年に刊行された「RユーザのためのRStudio[実践]入門~tidyverseによるモダンな分析フローの世界」の改訂2版である。初版は表紙のデザインから「#宇宙本」の愛称で呼ばれる。
「宇宙本」はタイトルに「Rユーザのため」とあるように幅広い対象読者を想定している。以下に各章の内容を箇条書きで示すが、これらの内容はどんな分野であっても役に立つスキルとなっている。
- Rの実行環境であり、伴侶となるRStudio
- データ入力とウェブデータの取り扱い
- データハンドリング
- 可視化
- レポート作成
なので、まずはR言語の初心者に本書をおすすめしたい。本書はR言語の入門書ではないので、オブジェクトの扱いであったりパッケージや関数の関係を伝えるものではない。また統計解析やプログラミングの細かな話も出てこない。しかし先に書いた通り、Rを使う上で役立つ内容がカバーされている。
特に読者にとって嬉しいのは扱われる項目がtidyverse中心となっている点である。tidyverseについて初心者は知らないと思うので本書から引用させてもらう。
本書では、tidyverseと呼ばれるパッケージ群を積極的に使います。tidyverseは、単なるパッケージの寄せ集めではなく、さまざまな操作を統一的なインターフェースで直感的に行える「tidyなツール群」を目指すものです。
tidyverseを扱わずに本書のような内容をまとめることはできるだろう。しかしtidyverseを使わなれば広い内容の解説が複雑になり、まとまりを欠いてしまうのは避けられないはずだ。tidyverseには確立されたマニフェストがある。本書はそれにならったパッケージを中心に扱っている。そのため関数や引数の指定に関して疑問を持つことが少ないのではないかと思う(少なくともわかりやすい関数名やパイプ演算子を使った処理を念頭に入れている点では)。
章の内容はもっと深く学べるものであるが、そこに入り込むと一冊の本になってしまう。特にデータハンドリングや可視化の作業は現実のデータ分析の場面にならないとわからないこともあると思う。しかしそうした現実問題においても本書の内容が役立つ機会があるはずだ。ゆえにRでの分析作業を進める上で必要な内容を一冊で学ぶことができる本書を勧めたい。
初版を持っている人へ
「宇宙本」の改訂版が出ると話を聞いて「なぜこの時期に?」というのが、初版を持っている私の最初の感想である。しかし理由はきちんとある。それはtidyverseの個々のパッケージに変更が加えられ、関数の追加・変更があったためだ。
本書のテーマとなっているRStudio、tidyverseの開発速度は速い。とりわけ、データハンドリングで欠かせないdplyr、tidyrパッケージのバージョンは初版ではそれぞれ0.7.4、0.7.2である。最新版はdplyr 1.0.6、tidyr 1.1.3なので、メジャーリリースである1.0.0を超えた点には注意である。また2章のウェブデータスクレイピングで取り上げられるrvestパッケージも1.0.0となっている。
メジャーリリースとなると、これまでのパッケージの使い方に対して大きな変化があることが多い。具体例をあげると、rvestパッケージは1.0.0となったことで html_node()、html_nodes()に取って代わる関数としてhtml_element()、html_elements()が提供された。また、dplyrでの複数列への処理で*_atや*_if()、*_all()を使うのは古いやり方で、今はacross()を利用するのが推奨されている。tidyrの縦長・横長にデータの形を変形させる関数 gather()や spread()は pivot_longer()、pivot_wider()に切り替わっている。本書はそうしたパッケージの更新を追うように加筆・修正が加えられている。当然、今後もこうした変更は入るだろう。しかし古い関数をいつまでも使っているわけにはいかない。書かれている内容として変わりはないようであるが、3年前の情報からアップデートし、2021年時点の情報(セーブポイント)として本書は重要なのだ。
また改訂2版では付録として
- stringrによる文字列データの処理
- lubridateによる日付・時刻データの処理
が追加されている。いずれもtidyverseのパッケージを扱っており、多くの場面で応用できる内容となっている。初版を読まれた方の中で、データ分析をしているときに文字列や日付・時刻データの処理に戸惑ったという人もいるのではないかと思う。3年前にはこれがなかった。なのでより実務の中で本書が活躍する機会が増えるのではないかと期待する。(欲を言えば、dplyrやggplot2の処理と合わせてこれらをどう使うのか、より応用的な解説があっても良いと思うが)
以上、内容が刷新され、より実務向きに参照できるようになったという理由で、初版をお持ちの方にも改訂2版をお勧めする。
なんとなくの物足りなさ
既存の内容についての変更は素晴らしいが、せっかくの改訂版なので、内容を追加されても良かったのでは?と思うところがいくつかある。これについては著者がそれぞれ悩まれていることだし外野が騒いでも仕方がないことだけど、一読者の感想として簡単に書いておく。
- 以前の関数名との比較 ... アップデートした関数は書かれているが、それが古い関数の何と対応しているのか書かれていない。それがないと過去のコードを見たときに比較、置き換えることができないのではと思う。
- 関数のlife cycle ... tidyverseのパッケージに含まれる関数の多くは、関数の開発状況に応じて"life cycle"と呼ばれるバッジをドキュメントに付与している。これを見ると、関数が安定して利用できるものなのか、廃止されたものなのかといった情報を得られる。本書が出た後でも読者がtidyverseパッケージの更新を追えるように、この情報の存在について書かれていても良かった。
- ウェブデータ取得でのhttrパッケージの存在 ... APIの例でhttrに触れずにrtweet、Seleniumの例だったのはなぜか。
- R 3.4.3から4.X.Y系へのアップデート ... R本体の内容は触れなくても良かったと思う一方、
data.frame(stringsAsFactors = )の挙動が変わった点は多くのユーザにとって影響が大きいと思う。これによって、組み込みのread.csv()では文字列の読み込みがデフォルトで因子型でなくなった。 - ggplot2パッケージの
geom_sf() - RStudioの作図デバイスバックエンドでのAGGの利用 ... RStudio 1.4.1103でも利用可能
- RStudioのPython連携
最後は少し愚痴っぽくなってしまった。お許しいただきたい。
日本語プロットの文字化けストレスを低減する - RStudio v1.4とraggパッケージを使う
RStudio v1.4とraggパッケージの登場でRStudio上で日本語の作図が面倒な指定不要で行えるようになりました。記事中で紹介する方法をとれば、RStudioのPlotsパネルに出力する図が文字化けしなくなります。RStudioユーザで日本語での作図を行う方にはぜひ知っていてもらいたいtipsです。
すごい!確かにグラフィックデバイスにAGGを指定すれば、ggplot2で日本語表示する際に面倒な ggplot2::theme_*(base_family = ) をしなくても、問題なく日本語が表示される。ハッピー( ^ω^ ) https://t.co/DL1ec5wj9U
— Uryu Shinya (@u_ribo) 2021年2月17日
なお、この記事の元ネタは https://www.tidyverse.org/blog/2021/02/modern-text-features/ で紹介されている通りです。詳細を知りたい方はぜひこちらのページや文末の参照ページ掲載のリンクをください。
RStudioと文字化け
みなさん大好きなRStudio、開発チームが頑張っていて、日本語を含めた多言語対応が進んでいます。一方で、日本語ユーザである我々にとっては、痒いところに手が届いていないと感じる部分や使い勝手の不自由さを感じる面もあります。具体的には「文字入力」と「文字化け」、これら2つの事柄はRStudioで日本語などのマルチバイト文字列を利用する際に問題となりやすい印象にあります。
文字化けは作図を行う際に生じます。何も考えずに次のコードをRStudioで実行すると、Plotsパネルに表示されるプロットにはmain引数に指定した文字列が正しく出力されず、いわゆるトーフ状態となります。
plot(mtcars$mpg, mtcars$disp, main = "排気量と燃費の関係")

これはR標準のグラフィックス機能の他、可視化パッケージとして有名なggplot2でも生じます(結果の出力は省略します)。
library(ggplot2) p <- ggplot(mtcars, aes(mpg, disp)) + geom_point() + labs(title = "排気量と燃費の関係") p
解決策はいずれも出力時に日本語の書体を指定することです。ここではmacOSで利用可能な日本語フォントの一種、「ヒラギノ明朝」のウェイトバリエーション「W3」を指すHiraginoSans-W3で日本語文字列を表示するようにプロットする例を示します。
par(family = "HiraginoSans-W3") plot(mtcars$mpg, mtcars$disp, main = "排気量と燃費の関係") p + theme_gray(base_family = "HiraginoSans-W3")
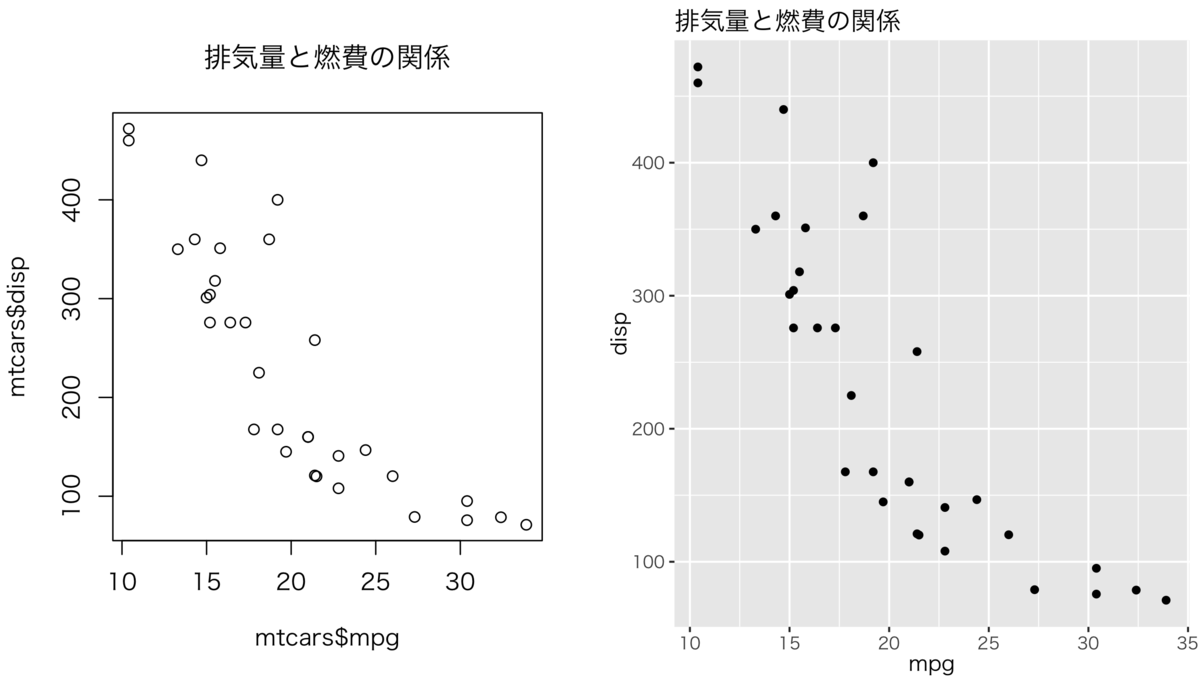
簡単なコードを追加するだけですが、毎回やるのは面倒です。またggplot2では既定の書体をtheme_set()を利用して次のように設定可能ですが、できればこの設定を意識せずに使えると楽ですよね。
こうして我々が悩まされてきた文字化けの問題がRStudioのバージョン1.4とraggパッケージによって改善される、というのが本題です。RStudio v1.4は2021年3月時点での最新版です。こちらからダウンロード可能です。また、raggはRパッケージを管理するCRANに登録されており、install.packages("ragg")を実行するとインストールされます。それでは実際にRStduio v1.4とraggを使って楽に日本語プロットを行うようにする設定を見ていきましょう。
RStudio v1.4とraggパッケージの組み合わせで文字化けを回避
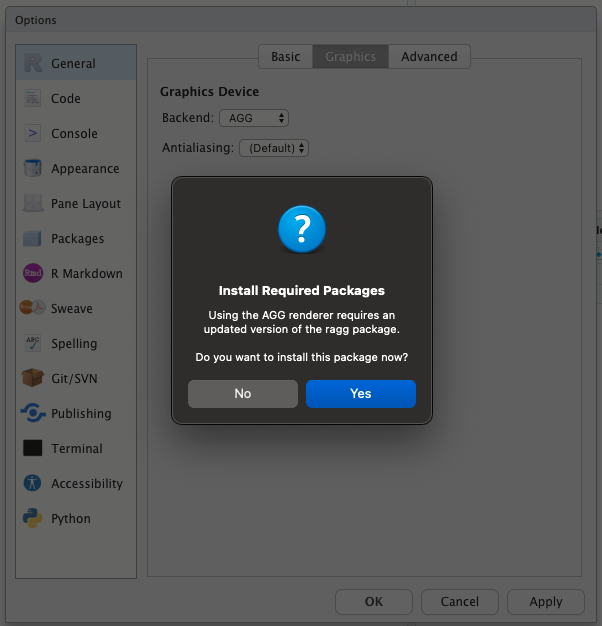
RStudio v1.4とraggパッケージの用意は済んでいますか?両方が揃ったら(raggの追加は後でも可能なので、最低限RStudioを導入してください)、RStudioの環境設定(Preferences)、General、Graphicsの中のGraphic Device、Backendの項目からAGGを適用するだけです。AGG(Anti-Grain Geometry)はraggパッケージが実装する作図デバイスのバックエンド処理で、この指定により日本語の文字化けが解消されます。一度設定すれば、RStduioを再起動しても設定は引き継がれます。
以下、細かい手順を解説します。RStudioの環境設定(Preferences)を開きます。環境設定を開くと、左側にGeneral、Code、Appearanceと並ぶ画面が表示されます。画面が表示された時の位置がGeneralです。そのまま右側のボックスに目を向けましょう。こちらにはBasic、Graphics、Advancedのタブがあり、現在はBasicの設定内容が表示されていると思います。ここでGraphicsのボタンをクリックして作図に関する設定を変更します。Graphic DeviceのBackendの項目が選択可能になっていますので、ここでAGGを選び、Applyボタンをクリック、OKボタンによって設定を反映させましょう。この時、もしraggパッケージがインストールされていない状態であれば、インストールを促すポップアップが表示されるので、まだの場合はここで行ってください。設定はこれだけです。
AGGを適用させたら、もう一度日本語のプロットを行ってみましょう。今度は文字化けせずに正しく日本語文字列が表示されているはずです。
注意点
上記の設定はあくまでRStduio上での作図(スクリーン上での表示)に機能します。そのため、ファイルやR Markdownでの出力の際には従来通りフォントの指定を行わないと文字化けします。ですので出力デバイスの指定にraggパッケージが提供する agg_*()の利用や、チャンクオプションでdev='ragg_png'を与えることをお勧めします。
ファイルへの出力
まずは標準の保存方法を示します。以下のコードで出力される図は日本語の文字化けがみられます。
# 日本語フォントの指定をしていないため、出力されたファイルで文字化けしている png("out.png") plot(mtcars$mpg, mtcars$disp, main = "排気量と燃費の関係") dev.off()
AGGを利用してpng形式で保存する際はragg::agg_png()を使います。ファイル名やサイズ、解像度などの指定は基本的にはpng()と同じです。
# 日本語フォントの指定不要で文字化けしない ragg::agg_png("out.png") plot(mtcars$mpg, mtcars$disp, main = "排気量と燃費の関係") dev.off()
ggplot2ベースでの保存、すなわちggsave()を使う場合にはさらに注意が必要です。デバイス指定の引数deviceにragg::agg_pngを与えた時に出力サイズが小さくなってしまう問題が報告されています。
これを防ぐためにheight、widthのオプションで出力サイズを大きくしようとすると次のエラーになります。
ggsave(plot = p, filename = "out.png", device = ragg::agg_png, height = 100 width = 100) #> Error: Dimensions exceed 50 inches (height and width are specified in 'in' not pixels). #> If you're sure you want a plot that big, use `limitsize = FALSE`.
この場合エラーメッセージの案内に従いlimitsize = FALSEを使いましょう。
もう一点、device=ragg::agg_pngを指定すると解像度のためのオプションggsave(dpi = )は機能せず、ragg::agg_png(res = )の値が適用されます。ですが次のようにしてggsave()の中で直接ragg::agg_png(res = )の値を操作可能です。
ggsave(plot = p, filename = "out.png", device = ragg::agg_png, # dpi = 320の代わりに指定 res = 320, height = 2240, # pixelsでのサイズ width = 2240, # pixelsでのサイズ limitsize = FALSE)
このあたりの処理はちょっとややこしいですが、問題解消のための機能改善が進んでいるようなので期待しましょう。
R Markdownでの利用
R Markdownでraggの機能を利用には、
- 図を出力するチャンクオプションに
dev='ragg_png'を指定する knitr::opts_chunk(dev = "ragg_png")を設定する- YAMLヘッダ部分に
dev: "ragg_png"を加える
のいずれかの方法をとります。チャンクオプションdev='ragg_png'を追加する場合は以下のようになります。
```{r, eval=FALSE, dev='ragg_png'}
# 文字化けしない
plot(mtcars$mpg,
mtcars$disp,
main = "排気量と燃費の関係")
```
![改訂2版 RユーザのためのRStudio[実践]入門〜tidyverseによるモダンな分析フローの世界 改訂2版 RユーザのためのRStudio[実践]入門〜tidyverseによるモダンな分析フローの世界](https://m.media-amazon.com/images/I/51dInR5rKdS._SL500_.jpg)