RStanのおさらいをしながら読む 岩波DS1 (ややアップデートした2018年版)
最近、以前書いた岩波DSの久保さんパートのおさらい記事を久保さん本人がRTしていたのを見て(ああ、あの頃はまだ大学院生をやっていたのだと懐かしくなったと同時に)、移り変わりのあるRコードは今も動くのか心配になった。結論を先に言うと、RPubsに書いたコードは現在の環境でも問題なく動作した。が、その頃にはtidyverseも十分に整備されていなかったし、私のR技術力も低く、ヘンテコなコードを書いていたのが気になる。そこで、新しいパッケージも出ていることだし、2018年版ということで再度実行してみた、というのがこの記事の顛末である。
元の記事でもstanコードは id:StatModeling さんの記事に書かれているものをコピーしたが、代入に「<-」を使うとstanが警告文を出すので「=」に書き換えておいた(非推奨なだけで実行自体はしてくれる。<-が非推奨〜云々は「StanとRでベイズ統計モデリング」でも書かれている)。

StanとRでベイズ統計モデリング (Wonderful R)
- 作者: 松浦健太郎,石田基広
- 出版社/メーカー: 共立出版
- 発売日: 2016/10/25
- メディア: 単行本
- この商品を含むブログ (9件) を見る
以下、変更した部分だけを書く。結果は変わらないので内容ではない。
まず例題として用意されているデータ(久保さんのウェブサイトからダウンロード)をdplyr::glimepse()で確認しているが、これと同じデータの確認は、私は最近skimrパッケージのskim()を使ってやることが多い。
skimr::skim(d) # Skim summary statistics # n obs: 20 # n variables: 6 # # Variable type: factor # variable missing complete n n_unique top_counts ordered # pref 0 20 20 10 A: 2, B: 2, C: 2, D: 2 FALSE # # Variable type: integer # variable missing complete n mean sd p0 p25 median p75 p100 hist # N 0 20 20 54.1 2.77 49 52.75 54 56 59 ▃▃▂▇▆▃▂▅ # X 0 20 20 0.5 0.51 0 0 0.5 1 1 ▇▁▁▁▁▁▁▇ # # Variable type: numeric # variable missing complete n mean sd p0 p25 median p75 p100 hist # Age 0 20 20 0.5 0.51 0 0 0.5 1 1 ▇▁▁▁▁▁▁▇ # mean.Y 0 20 20 155.3 2.8 151.36 153.06 155.69 157.22 161.71 ▃▇▁▂▇▁▁▁ # sd.Y 0 20 20 2.97 0.23 2.45 2.9 3.06 3.11 3.21 ▂▁▂▁▁▂▇▆
これだけでも数値からなる変数のデータのばらつきが把握できて大変良い。なおskimrパッケージの良さは id:niszet 氏が語ってくれている。
skimrはさらにここからdplyrパッケージの関数と組み合わせて利用することでグループ化したデータの要約や並びかえも行える。例えば、都道府県 pref 、測定回数 Age (1,2回目の測定をそれぞれ0と1で示す)ごとの平均身長 mean.Yの中央値を求め、平均身長の順に並びかえて表示するには次のようにする。dplyr(あるいはtidyverse)は、モデリングを行う際でも使う機会があるので最初に読み込んでおくと良いだろう。
library(dplyr) # skimr::skim()以外はすべてdplyrパッケージのデータ操作関数を利用 d %>% # 必要な変数にのみ制限 select(pref, Age, mean.Y) %>% # 都道府県、測定回数でグループ化し group_by(pref, Age) %>% # 統計量を求める skimr::skim() %>% ungroup() %>% # 統計量から中央値を抽出 filter(stat == "median") %>% # 平均身長の中央値が低い順に並びをかえる arrange(value) # # A tibble: 20 x 8 # pref Age variable type stat level value formatted # <fct> <dbl> <chr> <chr> <chr> <chr> <dbl> <chr> # 1 A 0 mean.Y numeric median .all 151 151.36 # 2 B 0 mean.Y numeric median .all 152 151.56 # 3 C 0 mean.Y numeric median .all 152 152.22 # 4 I 0 mean.Y numeric median .all 153 152.67 # 5 G 0 mean.Y numeric median .all 153 152.98 # 6 D 0 mean.Y numeric median .all 153 153.09 # 7 E 0 mean.Y numeric median .all 153 153.22 # 8 H 0 mean.Y numeric median .all 153 153.27 # 9 F 0 mean.Y numeric median .all 153 153.31 # 10 J 0 mean.Y numeric median .all 155 155.37 # 11 D 1.00 mean.Y numeric median .all 156 156 # 12 I 1.00 mean.Y numeric median .all 157 156.82 # 13 B 1.00 mean.Y numeric median .all 157 156.83 # 14 C 1.00 mean.Y numeric median .all 157 157.08 # 15 F 1.00 mean.Y numeric median .all 157 157.22 # 16 E 1.00 mean.Y numeric median .all 157 157.24 # 17 A 1.00 mean.Y numeric median .all 157 157.27 # 18 G 1.00 mean.Y numeric median .all 158 157.81 # 19 H 1.00 mean.Y numeric median .all 159 158.95 # 20 J 1.00 mean.Y numeric median .all 162 161.71
glm()の部分は依然としてbroomパッケージを使っているので変更なし。
さて以前はstanコードのdataブロックに渡すデータをリストで作成したが、ここも変わりがない。強いて言うなら、 list()の代わりにtibble::lst()にすることや、ユニークな都道府県の数をカウントした値(length(unique(d$pref)))はよく使うので、別なオブジェクトにしておくと良いだろう。また、length(unique(x))の部分はn_distinct(x)に書き換えるのが新しいやり方。もしくはこうした定数は別なファイルに記述して、configパッケージで必要な時に参照する方法もある。
n_pref <- n_distinct(d$pref) # list tibble::lst( N_r = 2, N_pref = n_pref, Mean_Y = t(matrix(d$mean.Y, n_pref, 2)), Sigma_Y = t(matrix(d$sd.Y / sqrt(d$N), n_pref, 2)), X = t(matrix(d$X, n_pref, 2)), Age = t(matrix(d$Age, n_pref, 2)))
stanコードのビルド、MCMCサンプリングの実行も変わりがない。記事を書いた時からStanは色々なアップデートがあっただろうが、動いてよかった..。
最後にサンプリング結果から推定したパラメータの可視化を行なっているが、ここもそのままのコードが動作する。ただしgridExtraを使った複数のggplot2作図の結合はちょっと古いかつ、若干わかりにくいのでeggあるいはpatchwork (2018年2月現在 GitHub版のみ利用可能 https://github.com/thomasp85/patchwork) パッケージで代替する。eggの方はgridExtraの開発者が中心に開発していて正式な後継っぽいのだが、個人的にはpatchworkの方が好み。
事後分布のヒストグラムやら、ggmcmcで作図するところまでは同じ。まずはeggを使った方法から。
library(egg) (p <- ggarrange(p1, p2, p3, p4, widths = 1:2))
library(patchwork) (p <- p1 + p2 + p3 + p4 + plot_layout(ncol = 2))
# egg, patchwork いずれも保存は同じ方法 ggsave(filename = "samping_parameters_result.png", p, width = 6, height = 4)

ああ、まだ「ベイズモデリングの世界」が買ったまま読めていない...。
他にも、私が追えていないだけで更新できる内容があるかもしれないが、2018年版ということでひとまずは Enjoy!

- 作者: 伊庭幸人
- 出版社/メーカー: 岩波書店
- 発売日: 2018/01/18
- メディア: 単行本(ソフトカバー)
- この商品を含むブログ (6件) を見る
![Rで楽しむベイズ統計入門[しくみから理解するベイズ推定の基礎] (Data Science Library) Rで楽しむベイズ統計入門[しくみから理解するベイズ推定の基礎] (Data Science Library)](https://images-fe.ssl-images-amazon.com/images/I/51844dFl3AL._SL160_.jpg)
Rで楽しむベイズ統計入門[しくみから理解するベイズ推定の基礎] (Data Science Library)
- 作者: 奥村晴彦,瓜生真也,牧山幸史,石田基広
- 出版社/メーカー: 技術評論社
- 発売日: 2018/01/16
- メディア: 大型本
- この商品を含むブログ (4件) を見る
(こっちもよろしくね😼)
hereパッケージの導入でファイル参照のパス問題の悩みを解消
去年から気になっていたものの、その利点や使い道について理解できていなかったhereパッケージ、ようやくにして少しわかった気がする。今は声を大にして言える。hereは良いぞ。hereをプロジェクトに導入することで、これまでにあったWindows - macOS間でのパス表記の違いや作業ディレクトリの階層性によるパスが正しく指定できないといった課題を解決することができるかもしれない。と言う訳で布教用の記事を書く。また、テスト用のリポジトリも用意しているので、hereを使ってそのご利益を享受されたい方はこちらをcloneなりダウンロードするなりして手前で実行してほしい。
参考) https://github.com/jennybc/here_here
問題
Rを使っていて、ファイルの保存先を指定するのに失敗してイラつきを覚える、そんな経験は誰しもがあるだろう。Rでは、作業ディレクトリ (working directory。getwd()で現在の作業ディレクトリを確認する)と呼ばれる基盤となるディレクトリが設定される。これにより目的のファイルまで簡単にたどり着くことができるなどの利点もあるのだが、複数のプロジェクトを運用している場合、プロジェクトのフォルダをまたいでしまったり、作業ディレクトリを頻繁に切り替える作業が発生してしまう(setwd()により作業ディレクトリは変更される)。皆さんには作図したファイルの保存先にfigフォルダを作っていたのに、全く異なる場所にファイルを保存してしまった、ということがないだろうか?私はしばしばそんな経験をした。ここに書いたにもパスに関する多様な問題があるだろうが、もっとも頻繁に遭遇する問題だと思われる2点について、次にまとめる。
RStudioの一機能であるRプロジェクトは、プロジェクトに応じた作業ディレクトリの構築を可能にする。Rプロジェクトであることを示す .Rprojファイルはプロジェクトとして扱うフォルダのトップの階層に置かれ、そこを起点とした作業ディレクトリでプロジェクトを回していくことができる。プロジェクトに関する全てのファイルをプロジェクトの中に置くことで利便性が増し、上記の問題を防ぐことが可能となる。
一方で、ファイルの参照形式はOS間で異なることがあり、参照がうまくできないことがしばしばある。次のコードはWindowsにおいてdataフォルダ中のiris.csvを読み込むための処理だが、これをUNIXベースのOSで実行するとエラーとなる。UNIXではフォルダの区切り文字に/を使うのが習慣であり、他方Windowsでは区切り文字に/の他、\\ (¥)の使用が認められるためである。
# Windowsで実行可能な読み込みはUNIX環境ではエラーとなる read.csv("data\\iris.csv")
またプロジェクトの外部で、ドライブからパスを指定しなくてはいけない時も、Windowsでは次のように指定する。
# ドライブからファイルを参照する read.csv("C:/documents/uribo/here_demonstration/data/iris.csv")
UNIXではユーザディレクトリは /home/<username>であったり、/Users/<username>となっていたりするがホームディレクトリは ~で表記を省略することもできる。また省略されたパスをpath.expand()により完全な形で示せる。
# ホームディレクトリからファイルを参照する read.csv("~/here_demonstration/data/iris.csv")
path.expand("~/here_demonstration/data/iris.csv")
## [1] "/home/rstudio/here_demonstration/data/iris.csv"
これはプロジェクト機能をもってしても残り続ける課題である。
またRmdファイルをプロジェクト内のフォルダに保存しておくと、プロジェクトの作業ディレクトリよりも深い階層でRコードを実行することになるため、コンソールでの実行とRmdファイル中のコードの記述が異ってしまう。具体的には、data/iris.csvを参照するには、一つ階層を遡って../data/iris.csvとしなくてはいけない。また、Rmdファイルをより深い階層においた場合、さらに遡って../../data/iris.csvと記述することとなる。これに対応するのは手間である。
hereによるファイル参照
こうした課題を解決するために、hereパッケージは優れた機能を提供する。hereはCRANNからインストールができる。この記事を書いた際のバージョンは0.1である。Windows環境にインストールした際、backportsパッケージの古いバージョンをインストールしておかないといけないとエラーになったので、参考のリンクを貼っておく。
library(here)
## here() starts at /home/rstudio/here_demonstration
hereパッケージを読み込むと、上記のメッセージが出力される。出力先のパスが基点を示している。Rプロジェクトが同じ階層内にある場合、その階層が基点にみなされる。
hereの考え方としては、あるディレクトリをプロジェクトとみなした場合、ファイルの参照を常にプロジェクトの基点から表記する、というものである。関数hereはパスを表現するのに用いられるが、従来の区切り文字で表記する方法の他にRの文字列としてフォルダ名を与えていくことでパスを表現することもができる。
# 従来のパス表記方法 here("data/iris.csv")
## [1] "/home/rstudio/here_demonstration/data/iris.csv"
# 文字区切りでフォルダ、ファイルを示す here("data", "iris.csv")
## [1] "/home/rstudio/here_demonstration/data/iris.csv"
パスを出力するだけなので、ファイルの有無は気にしない。
file.exists("aaa/bbb/ccc.csv")
## [1] FALSE
here("aaa", "bbb", "ccc.csv")
## [1] "/home/rstudio/here_demonstration/aaa/bbb/ccc.csv"
c()を使ってこんなこともできる。複数の文字列を指定した際は各要素の位置に対応したパスが出力される。
here("data", c("aa", "bb"), "iris.csv")
## [1] "/home/rstudio/here_demonstration/data/aa/iris.csv"
## [2] "/home/rstudio/here_demonstration/data/bb/iris.csv"
here("data", c("aa", "bb"), c("iris.csv", "mtcars.csv"))
## [1] "/home/rstudio/here_demonstration/data/aa/iris.csv"
## [2] "/home/rstudio/here_demonstration/data/bb/mtcars.csv"
here()がその効力を発揮するのは、特に深い階層にあるRコードやRmdファイルを実行する時である。サンプルリポジトリに作成したhoge/fuga/sample2.Rmdからdata/iris.csvを参照するには../../data/iris.csvと記述しなくてはいけなかったところをhere("data", "iris.csv")の記述に置き換えられる。data/iris.csvを参照するには、常にhere("data", "iris.csv")とすれば良いのである。
dim(read.csv(here("data/iris.csv")))
## [1] 150 6
# 作業ディレクトリを変更しても data/iris.csvの参照方法は変わらない setwd("hoge/fuga/") # The working directory was changed to /home/rstudio/here_demonstration/hoge/fuga inside a notebook chunk. The working directory will be reset when the chunk is finished running. Use the knitr root.dir option in the setup chunk to change the working directory for notebook chunks. dim(read.csv(here("data/iris.csv")))
## [1] 150 6
# 作業ディレクトリの変更は維持される getwd()
## [1] "/home/rstudio/here_demonstration/hoge/fuga"
なお、RMarkdownのチャンクでは、チャンク内で完結し、チャンクが切り替わると基点に戻る。
getwd()
## [1] "/home/rstudio/here_demonstration"
素晴らしい!
Rプロジェクトの外部でhere!
hereはRプロジェクトのあるフォルダを基点とするのが基本だが、他にも基点となり得るファイルやフォルダが認められている。GitリポジトリやRパッケージ開発に必要なDESCRIPTIONファイル(^Package:で始まること)などである。加えて、独自の.hereファイルがあるフォルダも基点にすることができる。これはRプロジェクトを用いない分析プロジェクトでは有効だろう。任意の作業ディレクトリでset_here()を実行あるいはpath引数にパスを指定して、.hereを作成できる。
パス問題は、これまで多く悩まされてきたものなので、Rプロジェクトの台頭によって大分ストレスがなくなったが、hereを導入することでさらにストレスフリーで作業を進められそうである。
Enjoy!
Rから離れたくない人向けのDocker環境の操作: RStudio Serverを分析・開発の基盤にするために
この記事はRStudioアドベントカレンダーの21日目の記事です。もうすぐこのアドベントカレンダーも終わりですね。ハヤイ!
今年のはじめにこんな記事を書きました。
皆さんはDockerを利用していますでしょうか。今年のデータ分析系のアドベントカレンダーでもぞうさんがdockerが取り上げられています。
Rユーザの自分にとっては、Dirk EddelbuettelやCarl Boettigerなどが携わるrockerプロジェクトが整備されているのが嬉しいです。
rockerプロジェクトのdockerイメージの多くはRStudio Serverをイメージのベースとしており、お手軽にローカル環境とは別のRStudio環境が構築できます。また必要に応じて、rockerのdockerimageをベースに俺俺dockerimageへ拡張するのも簡単です。俺俺dockerimageの必要性についてはTokyo.RのLTで発表を行った過去の資料もあります。
そんな便利なdockerですが、dockerコマンドを覚えられなかったりコマンドラインから実行するのが面倒だったりします。特に自分は常にRStudioの画面を開いていたい側の人間なのでRからdockerコンテナやイメージの操作ができると良いです。良いです(大切なことなので二回言いました)。
... というわけでdockerパッケージを使います。これにより、Rから直接dockerのあれこれが行えるようになります(実態はPythonのdockerモジュールをreticulateパッケージでラップしているだけ)。大好きなRからできるので、dockerで何ができるかも理解しやすくて良いですね。
dockerパッケージ
以下の説明はdockerが利用できる環境で、dockerモジュールがインストールされていることを想定しています。あ、Rのdockerパッケージのインストールも忘れずに。CRANからインストールできます。まずはdockerを起動した状態で次のコードを実行します。Rスクリプトと一緒にコマンドラインで実行する際のdockerコマンドも示します。
# install.packages("docker") library(magrittr) library(docker) client <- docker$from_env()
インストールされているイメージ一覧を出力します。
# docker images client$images$list() # [[1]] # <Image: 'uribo/tiny_rocker_geospatial:latest'> # # [[2]] # <Image: 'rocker/tidyverse:latest'> # # [[3]] # <Image: 'kaggle/rstats:latest'> # # [[4]] # <Image: 'rocker/rstudio:latest'> # # [[5]] # <Image: 'rocker/r-base:latest'>
Dockerイメージをpullしてくるにはpull関数を使います。
# docker pull uribo/tiny_rocker_geospatial client$images$pull("uribo/tiny_rocker_geospatial")
利用するdockerイメージの用意ができたらコンテナを起動しましょう。次のRスクリプトの実行は、docker run --rm -p 8787:8787 uribo/tiny_rocker_geospatial と同じです。
rss_instance <- client$containers$run(image = "uribo/tiny_rocker_minimum", remove = TRUE, # RStudio上で操作を続ける際はTRUEにする detach = TRUE, ports = list("8787/tcp" = "8787")) rss_instance$start()
これでコンテナが起動しました。localhost:8787をブラウザで開きましょう。また次のコードでコンテナの一覧を確認してみると、きちんとコンテナが動いていることがわかります。
# docker ps -a client$containers$list() # [[1]] # <Container: a94576e6a2>
コンテナを終了するにはstop関数を使います。現在のコンテナはremoveオプションを有効にしていたため、コンテナを停止すると自動的にコンテナが削除されます。
# docker stop <コンテナID> rss_instance$stop() # 停止とともにコンテナは削除される # docker ps -a client$containers$list() # list
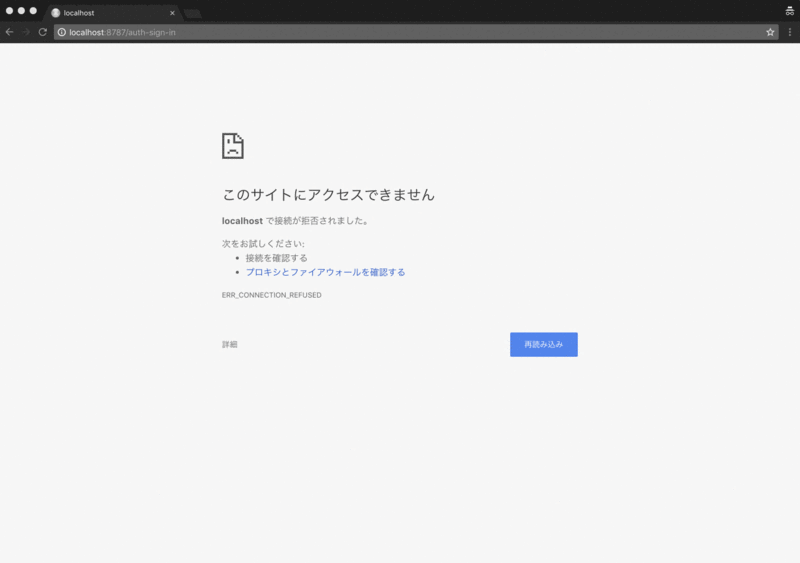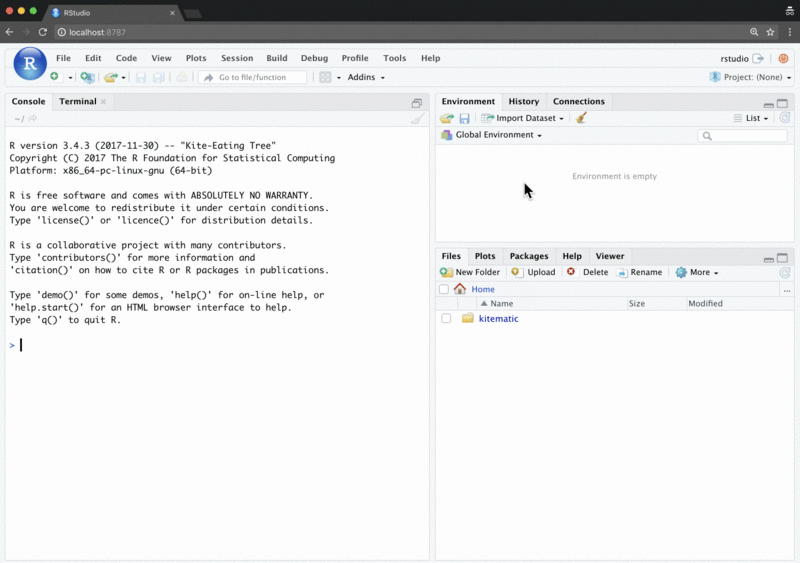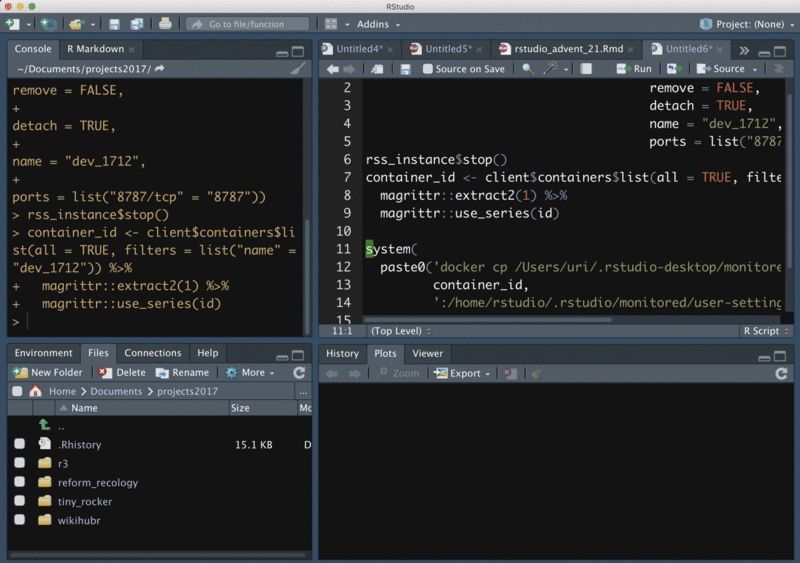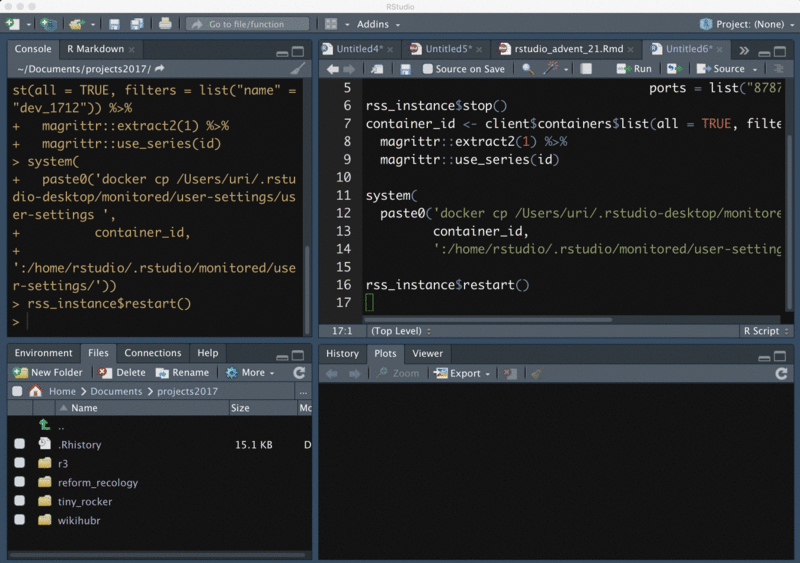
基本はこれでOKです。既存のコンテナを起動するには、まずコンテナのIDを知る必要があります。あるいはコンテナに名前をつけている場合にはそれを使うことも可能です。次はコンテナに名前をつけ、停止しても再起動できるように永続化させましょう。
rss_instance <- client$containers$run(image = "uribo/tiny_rocker_geospatial", remove = FALSE, detach = TRUE, name = "dev_1712", ports = list("8787/tcp" = "8787")) # 再起動を行うため停止します rss_instance$stop()
では停止しているコンテナを再起動します。再起動の方法には色々ありますが、ここではコンテナ名からコンテナidを抽出し、それを利用する方法を用います。
httr::http_error("http://localhost:8787") # Error in curl::curl_fetch_memory(url, handle = handle) : # Failed to connect to localhost port 8787: Connection refused
container_id <- client$containers$list(all = TRUE, filters = list("name" = "dev_1712")) %>% magrittr::extract2(1) %>% magrittr::use_series(id) cll <- docker$APIClient() cll$start(resource_id = container_id) httr::http_error("http://localhost:8787") # [1] FALSE
APIClient関数からcllというオブジェクトを作成し、コンテナを起動しました。dockerパッケージは便利ですが、コンテナの再起動をできないとRStudioを終了できないので困りますが、これで安心です。
明示的にコンテナを削除するには次のようにします。
cll$stop(resource_id = container_id) # docker rm <コンテナ ID> cll$remove_container(resource_id = container_id)
前述の通りdockerパッケージはPythonのdockerモジュールをラップしているので、dockerパッケージで何ができるのかを知りたい時はドキュメントを読むのが早いです。
ローカル環境のRStudioの設定を適用する
ここからは応用編です。起動したRStudio Serverでは、当然ながらRStudioの初期設定が適用されています。これではコンテナを作る度にぽちぽちとマウス操作によって設定をしていく必要が生じるので、楽にやりたいです。
なので、ローカルの設定をRStudio Serverへコピーするという方法をとっています。
system( paste0('docker cp /Users/uri/.rstudio-desktop/monitored/user-settings/user-settings ', container_id, ':/home/rstudio/.rstudio/monitored/user-settings/'))
APIがあるかと思ったのですが、なかなかうまくいかず...まだ試行錯誤なのですがこの方法で一応ローカルで動かしているRStudioのテーマやパネルの配置等の設定をRStudio Serverへ持ってくることができます。
注意としては、一度対象のコンテナを起動していないとコピーが成功しない(.rstudioという隠しフォルダが作成されない)、パスは絶対パスでなければいけないの2点です。
マウント: ローカルのファイルと同期させる
docker環境で作業して、それをローカルに保存したり、ローカルのコードをdocker環境で試す、ということがあります。それにはvolumeオプションを利用します。これはコンテナの作成時にやっておきます。
まずはdockerコンテナに構築したいローカルのパスとdockerコンテナ内の関係をオブジェクトとして作成します。modeは書き込みと保存ができるようにrwとしましょう。
mount <- list("/Users/uri/Documents/projects2016/jpmesh" = list("bind" = "/home/rstudio/jpmesh", # rw: read and write (ro: read only) "mode" = "rw")) rss_instance <- client$containers$run(image = "uribo/tiny_rocker_geospatial", remove = TRUE, detach = TRUE, volumes = mount, name = "dev_1712", ports = list("8787/tcp" = "8787"))

ユーザ、パスワードを変更する
RStudio Serverでは初期ユーザ名とパスワードがrstudioになっていますが、運用していくためには変更したいです。これもコンテナの起動時に設定しておきます。ここではenvironmentオプションを使います。USER、PASSWORDという値をそれぞれ任意のユーザ、パスワードにすることが可能です。以下は、ユーザ、パスワードの変更とついでに管理者ユーザとなるオプションも有効にする例です(追加でシステムのインストールが必要となることがあるため)。
rss_instance <- client$containers$run(image = "uribo/tiny_rocker_geospatial", remove = TRUE, detach = TRUE, environment = list("USER" = "piyo", "PASSWORD" = "hogepass", # sudo が有効になる "ROOT" = "TRUE"), ports = list("8787/tcp" = "8787"))

俺俺dockerfileの作成方法についてはまたの機会に。ちなみに、この記事で使っているコンテナは俺俺イメージの一つです。地理空間データを扱う上で欠かせない存在となったsfパッケージのインストールが面倒なのでサクッとできるようにしています。
https://hub.docker.com/r/uribo/tiny_rocker_geospatial/
Enjoy 🐳